2018-12-01
基因编辑技术是指对目标基因进行编辑,实现对特定DNA片段的敲除、插入等。自CRISPR/Cas9基因编辑技术问世以来,取得了一系列重大突破,并相继在2012、2013、2015和2017年被Science杂志评为十大科学进展之一。因此,CRISPR/Cas9以其操作简便和成本低廉等优势受到了众多研究者和投资者的青睐。
如下图所示:从2011到2018年,NIH对CRISPR相关研究的资助从500多万美元快速增长到11亿美元,而且CRISPR相关科学出版物的数量也从最初的87篇急剧增长到3917篇,这些数据反映了CRISPR /Cas9的巨大潜在价值。
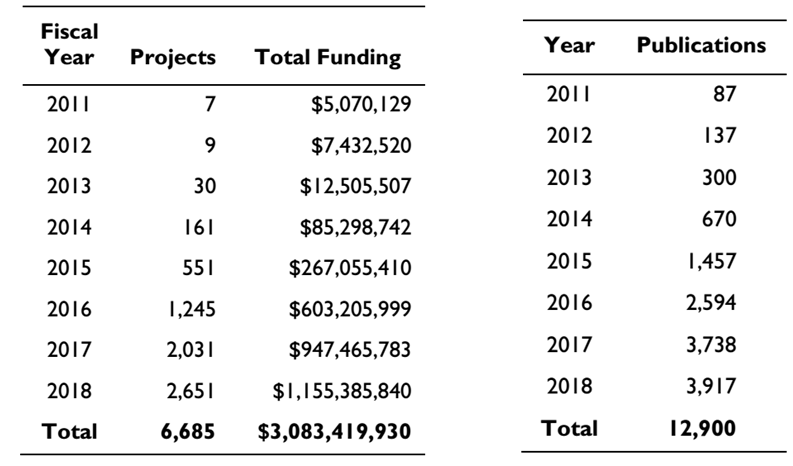
图1. 2011-2018年期间CRISPR研究经费(左)及出版物数量逐年上升(右)(来源NIH)
2018年,CRISPR系统继续发力,在多个领域取得突破性的进展,本期我们就先从CRISPR系统开发及机制研究方面来梳理一下相关的重大事件。
一、新型CRISPR系统继续拓展基因组编辑范围
自2013年最早公布的spCas9以来[1],科学家一直致力于在复杂的细菌群体中寻找更多Cas9的同系物来拓展基因编辑工具文库,以克服现有Cas9系统存在的诸多问题,比如组分太大无法包装入AAV(腺相关病毒载体)、PAM无法覆盖整个基因组等等。随着saCas9,Cpf1和Cas13等相继问世(如图),新型基因编辑系统被发现除了编辑DNA,逐渐开始具有编辑RNA以及单双链核苷酸等更多功能,这些新系统的发现不仅拓展了CRISPR的编辑范围,还延伸了其在诸多领域中的应用。2018年,又有更多有潜力的CRISPR系统相继被科学家们鉴定并改造。
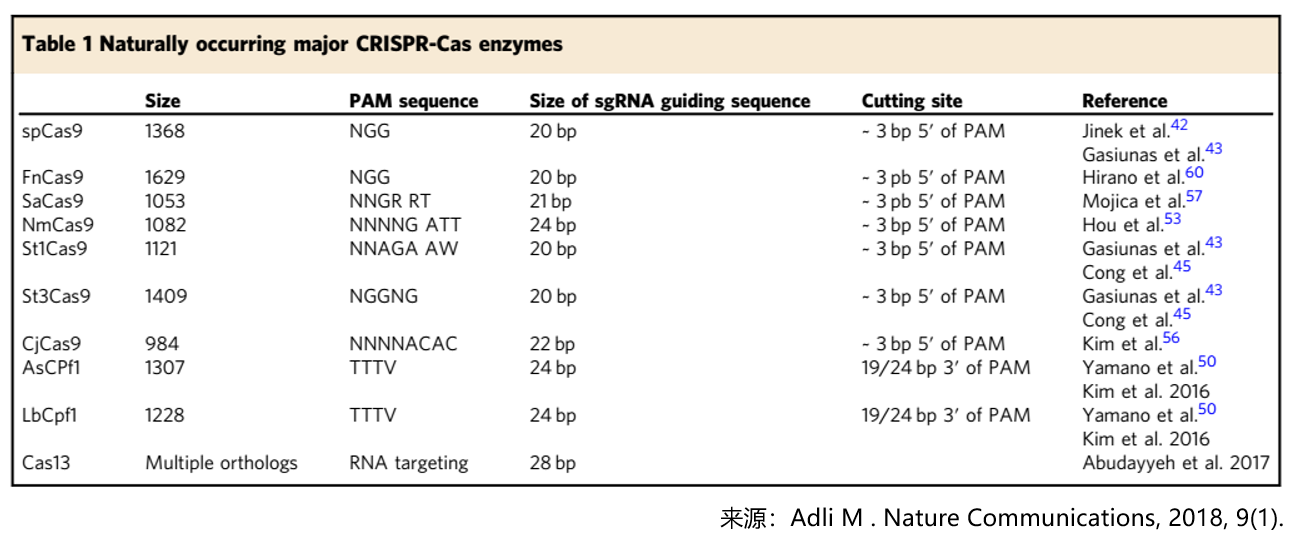
图2. 近年来被报道出的 Cas9同系物
1. CasRx:更强版本的Cas13系统
2017年底张锋团队首次报道发现Cas13系统,确认了其可以靶向哺乳动物细胞中的RNA[2]。
2018年3月15日,Salk研究所的Konermann等人为Cas13家族再添一名新成员:Cas13d。这是一种来自肠道细菌(黄化瘤胃球菌XPD3002)的CRISPR/Cas系统,被命名为CasRx。同Cas13类似,CasRx能够特异性的靶向并切割RNA,但比其他Cas13分子量小20%。同时CasRx介导的敲低效果相对于其他RNA调控方法具有更高的效率和特异性[3]。
2. 环状核酸酶(ring nuclease):病毒防御状态的终止“开关”
III型效应复合物最近被证明可结合入侵病毒的遗传物质形成一种环状寡腺苷酸,俗称第二信使,这一分子会通过CRISPR相关的Rossman折叠结构域结合并激活核糖核酸酶和其他因子来抵抗病毒的入侵,使细胞进入抗病毒状态。然而这种状态的持续激活对细胞是不利的,研究人员推测可能存在某种机制,在病毒被清除后关闭这种状态。2018年9月19日,Malcolm White团队证实了这一机制,一种被称为环状核酸酶的蛋白可特异性地切割环装寡腺苷酸,从而终止抗病毒状态。环状核酸酶的鉴定增加了人们对CRISPR系统的理解 [4]。
3. Cas14: 目前最小型Cas蛋白,为疾病诊断又添一利器
2018年10月18日,Jenifer Doudna团队发现了迄今为止最小的功能性CRISPR系统---Cas14上。Cas14只有400-700个氨基酸,但它同Cas12和Cas13一样,能够靶向切割单链DNA ( ssDNA ),且没有限制性序列要求,因而会盲目地切割细胞内所有的ssDNA。这一特性使得高保真单核苷酸多态性基因分型成为可能。通过进一步的改进,可以为目前已有的诊断系统(DETECTR)提供更多的选择[5]。
4. Cas12蛋白新组员,进一步拓展CRISPR系统的工具箱
Cas12a(Cpf1)同SpCas9已被作为最为常用的CRISPR-Cas基因编辑工具,并成功应用于基因组工程的各个领域。相比SpCas9,Cas12a以其较小的体积(1228bp)和仅需单个RNA的引导等优势被广泛研究和使用[6]。2018年12月6日来自美国Arbor Biotechnologies的研究人员发现了一组新的Cas12蛋白成员:Cas12c、Cas12g、Cas12h和Cas12i。其中的Cas12c、Cas12h和Cas12i具有RNA导向的双链DNA切割活性,还发现Cas12i在CRISPR的crRNA间隔区的互补链和非互补链上表现出明显的切割效率差异,这导致dsDNA形成的主要是单链切口(nicking)。Cas12g则主要以核糖核酸酶的形式,通过RNA靶向切割单链RNA和单链DNA。这项研究揭示了V型CRISPR-Cas系统在不同路线进化中的功能多样性,同时进一步扩展了CRISPR工具箱的运用范畴[7]。
二、工程化CRISPR系统拓展基因组应用范围
尽管CRISPR-Cas9系统被广泛用于基因组编辑,但Cas9可以识别的序列范围受到特定原间隔基相邻基序( PAM)需求的限制,因此通常很难实现高精度靶向双链DNA断裂的基因组编辑应用,这包括目前热门的单碱基编辑和基于CRISPR的基因筛选等技术。随着Cas9蛋白组学的深入研究,通过人为引入随机突变来改变PAM特异性的Cas9衍生物使突破这一限制成为可能。目前,科学家主要通过结构信息、基于定向进化的细菌选择系统来识别不同PAM序列的Cas9功能突变体,这些Cas9突变体具有与野生型SpCas9相当的编辑能力和特异性。这一技术为寻找高精度的Cas9突变体提供了研究方向,极大地拓展了CRISPR系统工具包,为实现CRISPR系统全基因组编辑打开了大门[8]。
1. 建立新型细菌防御系统筛选系统,开发更多具有潜在价值的分子工具
2018年1月25日,来自以色列魏茨曼科学研究所的Rotem Sorek团队在Science上发文,通过构建出一种扫描所有细菌基因组的计算机程序来研究更多的防御系统基因,将合成的多基因系统插入到天然免疫系统已被灭活的细菌中,通过噬菌体和其他感染因子的筛选,发现细菌存在10种之前未知的细菌免疫防御机制。这些新型防御系统中的任何一种都有可能成为下一种基因编辑工具 [9]。
2. xCas9: 一种能够识别多种PAM序列的SpCas9突变体
2018年2月28日,David Liu团队在Nature期刊上发文,他们利用一种噬菌体辅助的连续进化系统 ( PACE ) 进化出一种能够识别多种PAM序列的SpCas9变体( xCas9 )。xCas9的PAM相容性是迄今为止在所有Cas9识别范围的哺乳动物中最为广泛的,并可以应用于人类细胞中,包括靶向转录激活、核酸酶介导的基因敲除,单碱基编辑等。值得注意的是,尽管xCas9的PAM兼容性有所扩大,但它的DNA特异性要比SpCas9高得多,在NGG PAMs和非NGG PAMs靶位点中全基因组的脱靶效应也都比spCas9低得多。其中最佳的xCas9 PAMs为NGN,占约四分之一的人类基因组。xCas9的出现极大地扩展了CRISPR系统的DNA靶向范围,使CRISPR系统变得更加精确和灵活[10]。
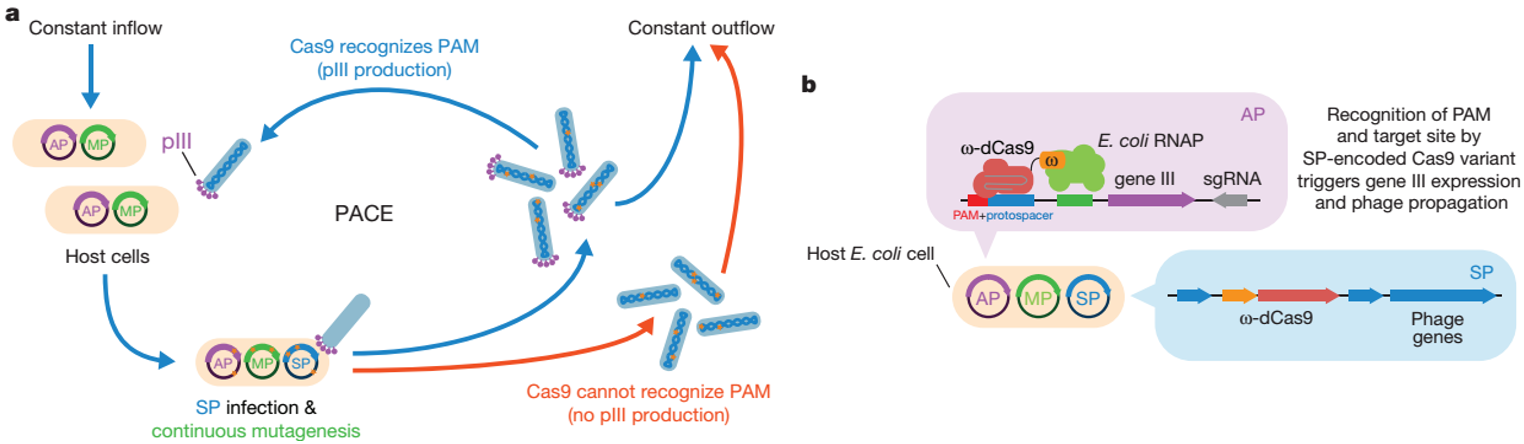
图3. PACE-噬菌体辅助连续进化器模式图[10]
3. SpCas9-NG:一种识别“NG”PAM序列的Cas9
2018年9月21日,Osamu Nureki团队通过合理的设计,开发了一种识别NG而不是NGG的SpCas9突变体(SpCas9-NG)。SpCas9-NG增加了靶向范围,且具有与野生型SpCas9相似的特异性,还可以与其他编辑器(胞苷脱氨酶)一起使用。因此,SpCas9-NG有效地补充了CRISPR工具箱,将在从基础研究到临床治疗等广泛应用中发挥重要的作用[11]。
4. ScCas9: 利用生物信息学发现的PAM仅含一个“G”的Cas9
2018年10月24日,来自美国麻省理工学院的研究人员构建了一个自动化的生物信息学管道,通过目标比对搜索PAMs ( SPAMALOT ),进一步探索了被忽略的Cas9链球菌直形菌株的微生物PAM多样性,发现了一种来自犬链球菌的Cas9(ScCas9),展示了其在细菌和人类细胞中的精确编辑能力。ScCas9的 PAM序列为5’-NNGTT-3’,且仅含一个碱基G,因此能靶向SpCas9不能的靶DNA序列且位点更多。ScCas9即可作为替代的基因组编辑工具,也可作为发现新的链球菌PAM特异性的功能平台[12]。
三、CRISPR系统核酸酶作用机制研究取得阶段性进展
Cas蛋白是CRISPR/Cas系统中的一类核酸酶,目前已鉴定出至少45个Cas蛋白家族。研究人员已通过冷冻电镜技术来解析其蛋白结构以及作用机制,为认识基因编辑系统的应用以及改造CRISPR系统提供了基本的结构和理论基础[13]。现在,各种新型Cas蛋白被陆续发现和解析,这将为开发新型CRISPR系统以及在将来高效地运用于基因治疗奠定基础。
1. 两篇研究揭示Cas4核酸酶对于CRIPSR免疫反应的重要性
2018年3月27日的,Shiimori团队在Cell Reports期刊上发文,证明了Cas4核酸酶是间隔序列前体临近基序( PAMs )靶向选择所必须的,有助于Cas1和Cas2选择新的CRISPR间隔物,并赋予天然宿主Synechocystis I- D CRISPR干扰能力[14]。同年,Shiimori团队于6月7日在Molecular Cell期刊上发表了工作,展示了Cas4在嗜热古菌P. furiosus中通过DNA序列整合的方式进行CRISPR-Cas免疫反应,并确定了两种Cas4(Cas4-1和Cas4-2 )在P. furiosus CRISPR间隔区DNA片段的获取及在原间隔区加工中的关键作用。另一方面,Cas4确保CRISPR间隔区在一个特定的方向上整合,最终触发CRISPR免疫反应[15]。总而言之,这些发现为Cas4核酸酶在CRISPR阵列的关键作用,为原间隔区生成和功能间隔区整合提供了体内依据。
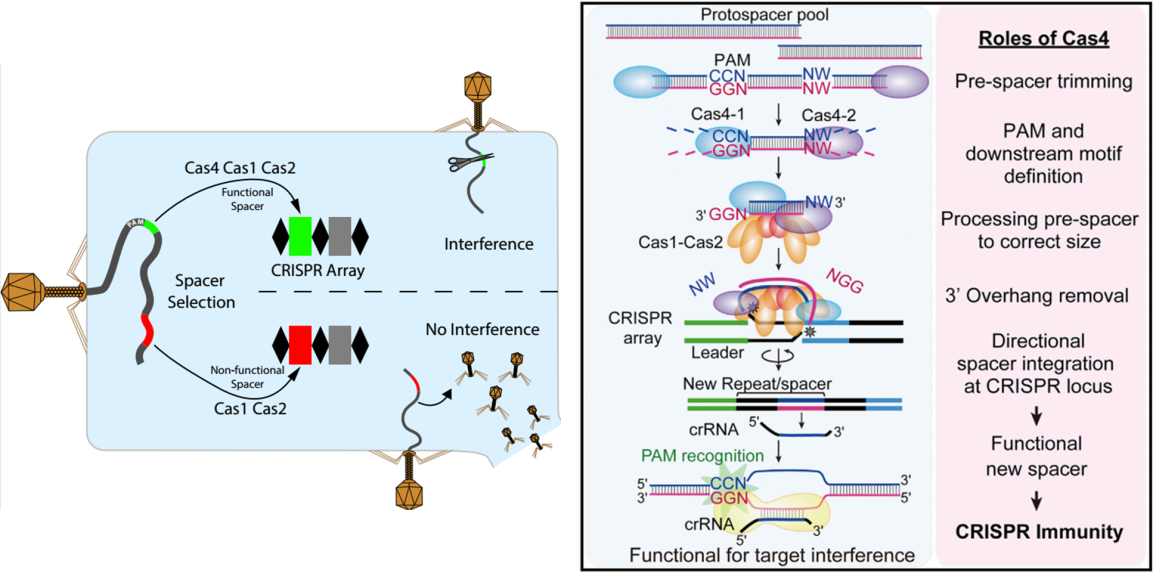
图4. Cas4的作用机制[14-15]
2. 冷冻电镜技术助力进一步解析CRISPR/Cas系统工作机理
尽管CRISPR系统已被广泛用于生物医学到生物技术或合成生物学等各个研究领域,但目前人们对其精细的工作机制仍然缺乏详细了解。今年三篇突破性的研究通过低温冷冻电镜技术分别揭示了不同CRISPR系统的结构以及工作机制,为CRISPR/Cas系统的改进及临床治疗提供了重要的理论基础。
1) I型CRISPR/Cas系统分子作用机制阐释:2018年7月6日,来自美国康奈尔大学和哈佛医学院的研究人员在Science期刊上发文,研究利用单颗粒低温电镜技术(cryo-EM)解析TfuCascade/R-loop/Cas3三元复合物在R-loop切割前后的结构,揭示了人们困惑的Cas3招募、DNA切割和降解机制。这些研究为理解I型CRISPR/Cas系统的分子作用机制提供了结构基础[16]。
2) Cas13d功能结构研究:2018年9月20日,美国Salk研究所的科学家们利用冷冻电镜解析了Cas13d-sgRNA二元复合物和Cas13d-sgRN-靶点RNA三元复合物的结构及一系列动态过程,这使得我们更详细地看到Cas13d-系统如何被引导到RNA并切割的过程。这为我们改进CRISPR系统,使其更为有效地为RNA的疾病治疗奠定基础[17]。
3) Cas12d构象循环解析:2018年12月13日,来自诺和诺德基金会中心的研究人员利用一种低温电镜技术,阐明了Cas12a靶向DNA切割和随意降解ssDNA的工作机制,揭示了Cas12a切割其靶DNA,释放非特异性切割活性,激活后降解ssDNA分子等工作机制。这使得我们可以通过调整CRISPR引擎来达到特定的预期效果[18]。
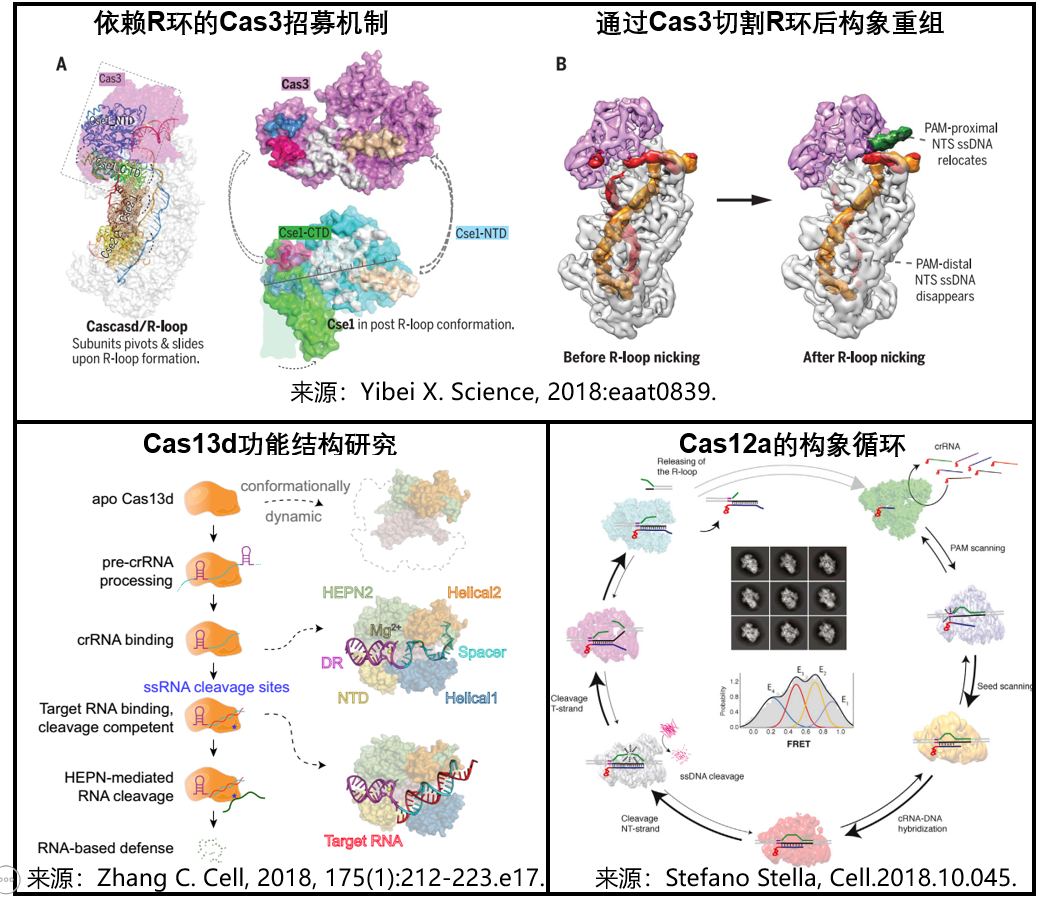
图5. 低温冷冻电镜技术揭示了CRISPR系统的结构以及工作机制[16-18]
四、基于CRISPR系统的基因编辑研究取得突破性进展
CRISPR/Cas基因编辑系统在人类治疗应用方面具有巨大的潜能,充分了解体内编辑系统的修复机制将有助于我们更加高效且准确地进行基因编辑,为安全有效的临床应用提供充足的理论和技术基础。
1. 范可尼贫血通路在CRISPR-Cas9编辑形成DSBs修复过程中发挥关键作用
2018年8月,美国加州大学伯克利分校的研究人员在Nature Genetics期刊上发表的研究工作,颠覆了之前的“细胞在Cas9酶剪切DNA后修复基因”的假说,研究人员通过CRISPR干扰技术对2000多个基因进行沉默,发现范可尼贫血通路(Fanconi anemia pathway)中的FANCD2蛋白会定位在Cas9诱导的DSBs上,表明它在调节基因组编辑中起着关键作用——调节FANCD2蛋白可以提高HDR频率。同时发现Fanconi贫血途径同NHEJ无关,而是通过提高同源重组效率使得修复机制转向单链模板修复机制,因而通过调控Fanconi贫血通路的活性可以提高HDR的编辑效率。这一发现将有助于提高在基因组中插入外源DNA的效率,并确保CRISPR编辑获得预期的结果[19]。
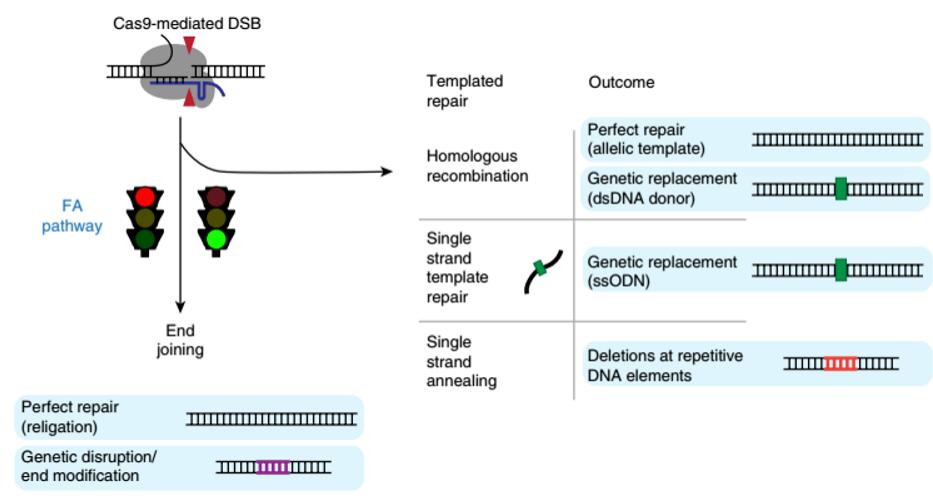
图6. 范科尼贫血( FA )途径介导的双链断裂( DSB )修复[19]
2. 精准且可预测的CRISPR染色体重排揭示Cas9介导的碱基插入规律
2018年7月19日,来自上海交通大学的吴强课题组在Molecular Cell期刊上发表了团队的最新研究成果,颠覆了现有的基因编辑理论。他们发现Cas9切割DNA产生的切口会产生突出末端,而不仅仅只有之前报道的平口末端的形式,同时利用高通量测序针对成对导向RNA编辑后DNA修复方式进行分析,发现断裂末端的修复方式是可预测的,呈现出非常有规律的形式。这一颠覆性的发现为优化和改造基因编辑技术奠定了坚实基础[20]。
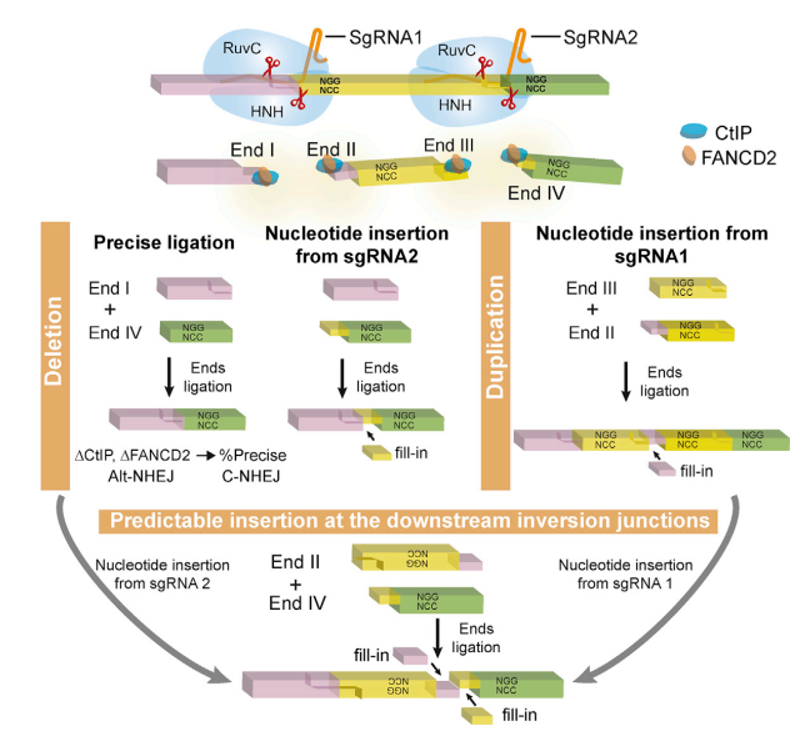
图7. CRISPR系统切割后的修复是精准且可预测的[20]
3. 无模板CRISPR/Cas9基因编辑的精准性被揭示
长期以来,在没有提供体模板的情况下,CRISPR切割后的修复通常被认为是随机且异质的。2018年11月7日在Nature期刊上HMax W. Shen团队发现,通过CRISPR/Cas9靶向基因组的2000多个位点来检测细胞是如何进行修复的,结果显示无模板的Cas9基因编辑是可预测的,经编辑的基因并不包含大量的变异,而是一种单一的结果。同时他们通过导入这些大数据,建立了一种机器学习模型(indelphi),它可以高精度地预测五个人类和小鼠细胞系中基因编辑的修复结果,并且将这一预测用于测试人类疾病的修复效果。这种模型能将致病基因型精确修复到预测的基因型,从而能够准确地校正人类疾病相关的突变。这项研究为精确、无模板的基因组编辑建立了基础[21]。
随后,2018年11月27日Felicity Allen团队在Nature 也发文,他们合成构建了超过4万多对导向RNA和靶向DNA序列的编辑文库系统探索CRISPR/Cas9的切割修复机制,明确了修复的结果是取决于靶向区域的DNA序列,大多数可再现的突变是单碱基插入、短的缺失或更长的微同源性介导的缺失的结果。同时也利用大数据开发了一种机器学习算法(FORECasT),能够实现仅使用靶位点DNA序列来预测纠正的结果[22]。这些机制的深入理解和新工具的开发将有助于人们更好地设计基因编辑实验。
综上,为大家简单介绍了2018年新型CRISPR系统的开发及机制研究方面的重要事件。目前CRISPR-Cas9已经成为一种简单、精确且快速的基因编辑技术,广泛应用于医学及农林牧等诸多生命科学相关领域,如医学中常涉及眼疾、血液病以及其他遗传病。同时我们也认识到,这一技术依然存在许多问题以及诸多潜力有待继续研究和拓展,我们相信在不久的将来CRISPR-Cas9将会在各个领域展现其不可估量的巨大价值。
参考文献:
1. 201302_Science_Multiplex Genome Engineering Using CRISPR-Cas Systems
2. Cox D B T , Gootenberg J S , Abudayyeh O O , et al. RNA editing with CRISPR-Cas13[J]. Science, 2017:eaaq0180.
3. Konermann, Silvana, et al. “Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors.” Cell (2018) pii: S0092-8674(18)30207-1.
4. Athukoralage J S , Rouillon C , Graham S , et al. Ring nucleases deactivate type III CRISPR ribonucleases by degrading cyclic oligoadenylate[J]. Nature, 2018.
5. Lucas B. Harrington1,*,†, David Burstein2,*,‡, Janice S. Chen et al. Programmed DNA destruction by miniature CRISPR-Cas14 enzymes. Science, 2018.
6. Zetsche B , Gootenberg J , Abudayyeh O , et al. Cpf1 Is a Single RNA-Guided Endonuclease of a Class 2 CRISPR-Cas System[J]. Cell, 2015:S0092867415012003.
7. Winston X. Yan et al. Functionally diverse type V CRISPR-Cas systems, 2018, doi:10.1126/science.aav7271.
8. Kleinstiver B P , Prew M S , Tsai S Q , et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities[J]. Nature, 2015, 523(7561):481-485.
9. Doron S , Melamed S , Ofir G , et al. Systematic discovery of antiphage defense systems in the microbial pangenome[J]. Science, 2018:eaar4120.
10. Hu J H , Miller S M , Geurts M H , et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity[J]. Nature.
11. Hiroshi Nishimasu, Xi Shi, Soh Ishiguro et al. Engineered CRISPR/Cas9 nuclease with expanded targeting space. Science, 21 Sep 2018, 361(6408):1259-1262, doi:10.1126/science.aas9129.
12. Pranam Chatterjee1,2,*,*,terjee1,2,*,1,2,*,ered CRISPR/Cas9 nuclease with expanded targeting space. Science, 21 Sep 20ortholog. Science Advances, 24 Oct 2018, 4(10):eaau0766.
13. Jinek M , Jiang F , Taylor D W , et al. Structures of Cas9 Endonucleases Reveal RNA-Mediated Conformational Activation[J]. Science, 2014, 343(6176):1247997.
14. Sebastian N. Kieper, Cristóbal Almendros, Juliane Behler et al. Cas4 Facilitates PAM-Compatible Spacer Selection during CRISPR Adaptation. Cell Reports, 27 March 2018, 22(13):3377–3384, doi:10.1016/j.celrep.2018.02.103.
15. Masami S , Garrett S C , Graveley B R , et al. Cas4 Nucleases Define the PAM, Length, and Orientation of DNA Fragments Integrated at CRISPR Loci[J]. Molecular Cell, 2018, 70(5):814-824.e6.
16. Yibei X , Min L , Dolan A E , et al. Structure basis for RNA-guided DNA degradation by Cascade and Cas3[J]. Science, 2018:eaat0839.
17. Zhang C , Konermann S , Brideau N J , et al. Structural Basis for the RNA-Guided Ribonuclease Activity of CRISPR-Cas13d[J]. Cell, 2018, 175(1):212-223.e17.
18. Stefano Stella,Pablo Mesa,Johannes Thomsen,et al. Conformational Activation Promotes CRISPR-Cas12a Catalysis and Resetting of the Endonuclease Activity, Cell (2018). DOI: 10.1016/j.cell.2018.10.045.
19. Chris D. Richardson, Katelynn R. Kazane, Sharon J. Feng et al. CRISPR–Cas9 genome editing in human cells occurs via the Fanconi anemia pathway. Nature Genetics, August 2018, 50(8):1132–1139, doi:10.1038/s41588-018-0174-0.
20. Precise and Predictable CRISPR Chromosomal Rearrangements Reveal Principles of Cas9-mediated Nucleotide Insertion
21. Max W. Shen et al., Predictable and precise template-free CRISPR editing of pathogenic variants, Nature, 2018, DOI: 10.1038/s41586-018-0686-x.
22. Felicity Allen et al, Predicting the mutations generated by repair of Cas9-induced double-strand breaks, Nature Biotechnology (2018). DOI: 10.1038/nbt.4317.